皆さんはDNAマイクロアレイ法と次世代シーケンス解析の違いについて説明できますか?また、実際に遺伝子発現を解析する場合に、どちらの方法が自分の目的に適しているか判断することができますか?
「それぞれの方法は講義で学んだが、違いがよく理解できなかった」「次世代シーケンス解析のほうが優れていると思うが、その理由は説明できない」「網羅的な遺伝子発現の解析を卒業研究で行うので復習したい」「自分の研究にどちらの解析方法が合っているか知りたい」
今回はこのような悩みを持つ学生に向けて、mRNAを網羅的な定量方法のDNAマイクロアレイ法と次世代シーケンス解析の一つのRNA-Seq(RNAシークエンス)、それぞれのメリットとデメリットやそれぞれを使い分けるポイントをまとめました。
遺伝子発現を網羅的に解析する目的
遺伝子発現を網羅的に解析する目的は、数多くの遺伝子の発現を一度に定量・解析することです。リアルタイムPCRやノーザンブロッティングでの遺伝子発現の解析は、一遺伝子ずつの測定と解析が必要です。そのため、複数の遺伝子発現を解析したい場合、解析した結果を研究者自身がまとめていました。しかしこれでは、多くの遺伝子発現を解析するには膨大な手間と時間が必要です。
そこで活用されるのが、網羅的な解析手法です。今回紹介・比較するDNAマイクロアレイ法と次世代シーケンス解析の一つであるRNA-seqは、遺伝子発現を網羅的に解析する方法です。
DNAマイクロアレイ法とは?
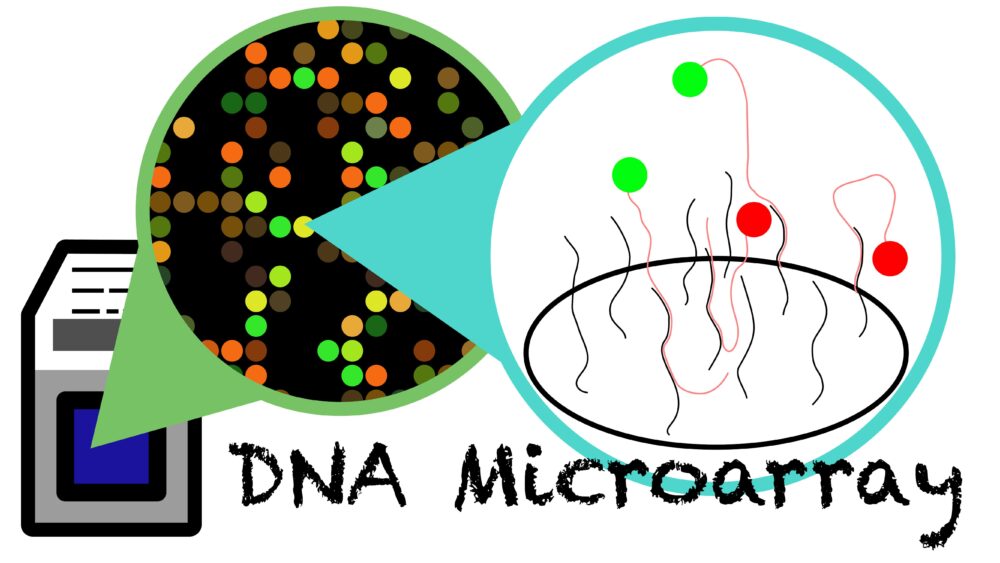
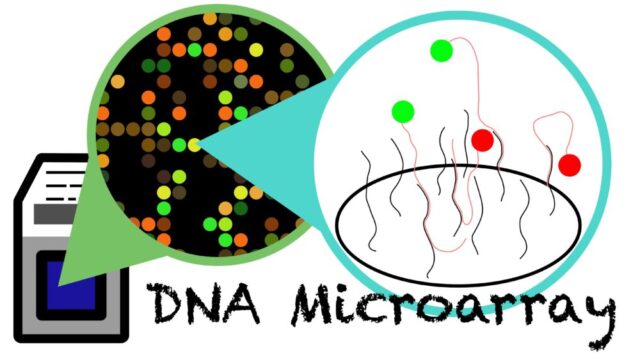
DNAマイクロアレイ法はDNAマイクロアレイ(別名、DNAチップ)を用いた遺伝子の網羅的解析の手法です。DNAマイクロアレイにはプラスチックまたはガラス製の基盤上に最大数万種類のDNA断片(DNAプローブ)が高密度に並べられています。サンプル遺伝子とDNAプローブのハイブリダイゼーションを検出して遺伝子発現を網羅的に解析します。
DNAマイクロアレイ法について詳しくはこちらの記事で解説しています。
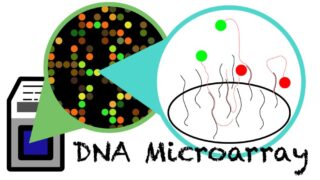
次世代シーケンス解析とは?
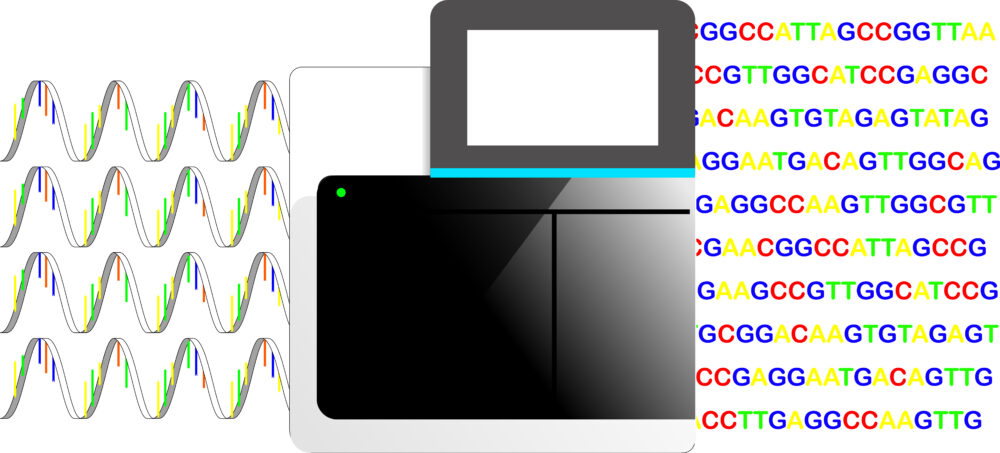

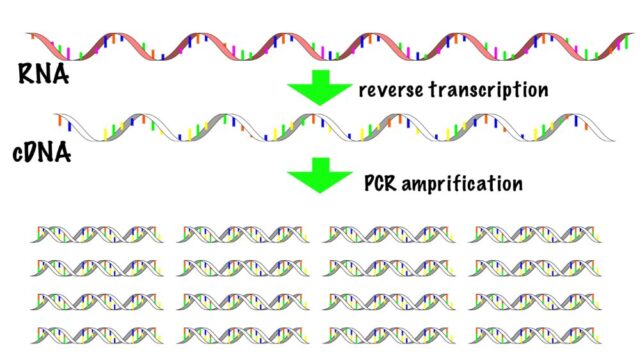
次世代シーケンス解析は、断片化した遺伝子の塩基配列を次世代シーケンサーで決定し解析する方法です。得られたデータからゲノム解析や遺伝子同士の相互関係の解析、タンパク質の構造や機能を予測します。
また、次世代シーケンス解析は次世代シーケンサーを用いた解析方法の総称です。そのため、解析する対象、目的によりいくつか種類があります。その一つがRNA-Seqです。RNA-SeqはmRNA発現量を網羅的に定量します。
RNA-Seqについては、詳しくはこちらの記事で解説しています。


DNAマイクロアレイ法とRNA-Seqの比較
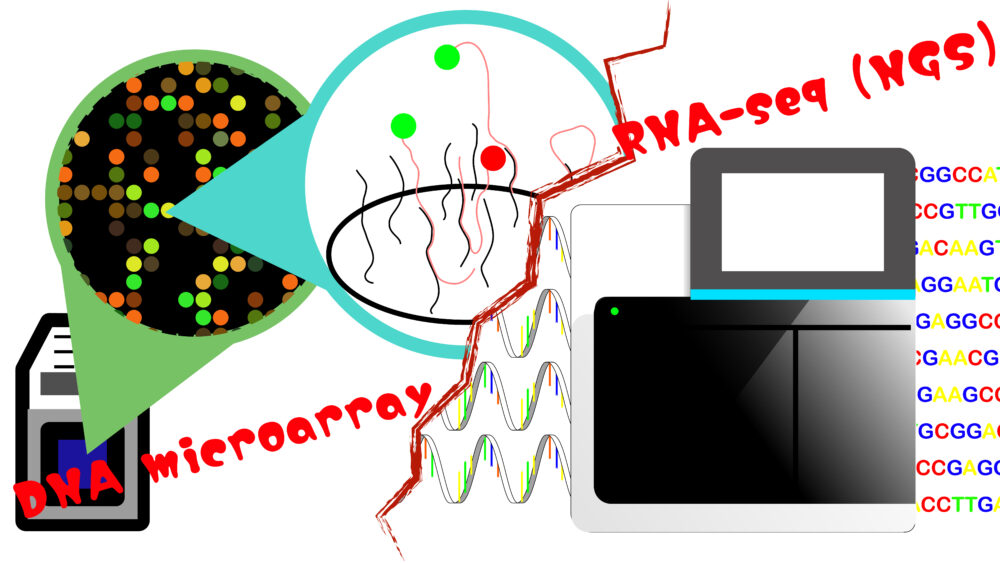

DNAマイクロアレイ法とRNA-SeqはいずれもmRNA発現を網羅的に定量できます。2010年代よりRNA-SeqがDNAマイクロアレイ法にかわり主流となりました。RNA-Seqは、サンプル全体のmRNA発現解析ができる点で、DNAマイクロアレイ法より優れていると考えられています。しかし、DNAマイクロアレイ法にもRNA-Seqにはないメリットがあります。そこで、DNAマイクロアレイ法とRNA-Seqそれぞれのメリットを解説します。
- データのサイズが小さい
- 短時間での解析が可能
- デ-タ解析の手法が簡単
- 塩基配列が未知のmRNAの定量ができる
- 発現量の少ないmRNA発現の定量ができる
- 得られる情報量が多い
それぞれのメリットを理解して、自分が行いたい網羅的解析にどちらの手法が適切か判断することが大切です。
DNAマイクロアレイ法のメリット
データのサイズが小さい
DNAマイクロアレイ法で扱うデータ量は、RNA-Seqの1,000分の1程度です。DNAマイクロアレイ法では、DNAマイクロアレイ上のDNAプローブと相補的な遺伝子発現しか検出できません。そのため、DNAマイクロアレイ法ではサンプル全体の遺伝子情報の一部しか得られません。その結果、扱うデータ量も少なく、データサイズはメガバイト単位です。データサイズが小さいため、保管や解析も、普段使用しているパソコンでも十分です。
一方RNA-Seqでは、1回の解析でサンプルの全ての塩基配列を決定します。そのため、得られる情報も多く、データサイズの単位はギガバイトです。データサイズが大きいため、データの保管や解析には専用のサーバーが必要でその分費用もかかります。
短時間での解析が可能
DNAマイクロアレイ法は、解析にかかる時間が短いです。DNAマイクロアレイ法で取り扱うデータサイズは、メガバイト単位です。そのため、解析ソフトウエアによっては数分で解析できます。
一方、RNA-Seqではギガバイト単位のデータを専用のサーバーを用いて解析する必要があります。そのため、解析には数週間必要になる場合もあります。
データ解析の手法が簡単
DNAマイクロアレイ法は、データの解析も比較的簡単です。DNAマイクロアレイ法で取り扱うデータサイズは小さく、短時間で簡単に解析できるソフトウエアも開発されています。
一方RNA-Seqを含め次世代シーケンス解析では、データの解析にバイオインフォマティクスの知識が必要です。バイオインフォマティクスとは生命科学と情報科学を合わせた領域です。次世代シーケンス解析では得られたデータからゲノム解析や遺伝子同士の相互関係の解析、タンパク質の構造や機能を予測します。次世代シーケンス解析では膨大なデータを処理する為に専用の解析ソフトウエアや解析されたデータを理解するための知識が必要になります。



RNA-Seqのメリット
塩基配列が未知のmRNAの定量ができる
RNA-Seqは塩基配列がわからないmRNAでも定量できます。RNA-Seqでは塩基配列を決定しながら解析します。そのため、塩基配列が未知のmRNAの定量、解析が可能です。
一方、DNAマイクロアレイ法での遺伝子発現の定量には、DNAマイクロアレイが必要です。DNAマイクロアレイの基盤上のDNAプローブを作成するためには、塩基配列が既知である必要があります。そのため、塩基配列が未知のmRNAは定量、解析できません。
発現量の少ないmRNAの定量ができる
次世代シーケンス解析は発現量の少ないmRNAも高感度に定量できます。RNA-Seqでは、塩基配列を決定しながらmRNA発現を解析します。そのため、発現量の少ない遺伝子でも高感度での定量が可能です。
一方、DNAマイクロアレイ法は発現量が少ないmRNAの定量感度は低いです。DNAマイクロアレイ法では、DNAプローブとのハイブリダイゼーションにより遺伝子発現を検出します。しかし、完全に配列が一致していないDNAプローブと偶然ハイブリダイゼーションすることがあります。そのため、データ解析の際は、一定よりも低いシグナルを無視するのが一般的です。その結果、微量な発現の遺伝子はデータを得ることが困難です。
得られる情報量が多い
RNA-SeqではDNAマイクロアレイ法に比べて多くの情報が手に入ります。RNA-Seqでは、 mRNAの塩基配列を決定しながら解析を進めます。そのため、もともと目的としていなかった未知の遺伝子情報が手に入る可能性があります。得られる情報量が非常に多い点がRNA-Seqのメリットです。
一方、DNAマイクロアレイ法ではDNAプローブと相補的な遺伝子の情報しか手に入らず、RNA-Seqと比較して限られた情報のみになります。また、未知の遺伝子の情報も得られません。



DNAマイクロアレイ法とRNA-Seqを使い分ける3つのポイント
DNAマイクロアレイ法とRNA-Seqのどちらが適切か確認するポイントは3つあります。
- 目的のmRNAの塩基配列はわかっているか?
- サンプルや遺伝子の発現量は十分か?
- サンプルの遺伝子情報の全てが必要か?
DNAマイクロアレイ法とRNA-Seqを比較すると、RNA-Seqのほうが一度の解析で得られる情報が多いです。しかし、測定したい遺伝子によってはRNA-Seqではオーバースペックとなります。それぞれの方法の利点・欠点から、自分の求めるデータがどちらの解析が適切かを確認することが大切です。
DNAマイクロアレイ法が良い場合
DNAマイクロアレイ法のほうが良いのは以下の条件を満たす遺伝子の解析です。キーワードは検索(既知の遺伝子を定量する研究)です。
- 目的のmRNAの塩基配列が既知
- mRNAの発現が十分量ある
- 未知の遺伝子情報を必要としない(配列既知の遺伝子情報だけでよい)
DNAマイクロアレイ法では、使用するDNAマイクロアレイのDNAプローブに相補的なmRNAしか定量できません。DNAマイクロアレイで定量できる遺伝子のみで十分な研究デザインであれば、RNA-Seqを用いるメリットは小さいです。
また、サンプル量が十分にあり、特定のmRNAの発現や定量が目的であれば、DNAマイクロアレイ法が適当です。この研究では、塩基配列がわかっている特定のmRNAの定量が目的です。そのため、DNAプローブを用いてmRNAを定量するDNAマイクロアレイを用いた解析で、目的は達成できます。また、未知の遺伝子情報や塩基配列を検索する必要がありません。このような研究ではDNAマイクロアレイ法がRNA-Seqで必要になるデータ解析の時間やサーバーなどへの金銭コストが小さい点で優れています。



RNA-Seqが良い場合
RNA-Seqを用いたほうが良いのは、以下の条件が必要な実験です。キーワードは探索(未知の遺伝子を含めて定量する研究)です。
- 塩基配列が未知のmRNAも含めて定量したい
- mRNAの発現量が少ない(または未知)
- 未知の遺伝子情報を探索したい
RNA-Seqでは遺伝子の塩基配列を決定して解析を行います。そのため、サンプルに含まれる全ての塩基配列、定量が可能です。したがって、RNA-Seqは塩基配列が未知のmRNAを含め網羅的な定量が必要な実験が適当です。
例えば、正常細胞とがん細胞のmRNAの違いについて網羅的に定量する研究を考えます。塩基配列が既知の遺伝子のみを測定対象とする場合は、DNAマイクロアレイ法でも定量できます。しかし、塩基配列が未知のmRNAや、未知のバリアントも含めて定量するような研究ではRNA-Seqが必要です。



まとめ
今回はDNAマイクロアレイ法とRNA-Seq、それぞれのメリットを解説しました。
数多くの遺伝子の発現を一度に定量、解析する
DNAマイクロアレイ(別名、DNAチップ)を用いて遺伝子を網羅的に解析する手法
断片化した遺伝子の塩基配列を次世代シーケンサーで決定し解析する方法の総称。その中で、mRNA発現量を網羅的に定量する方法がRNA-Seq
DNAマイクロアレイ法とRNA-Seqの比較
DNAマイクロアレイ法のメリット
- データのサイズが小さい
- 短時間での解析が可能
- デ-タ解析の手法が簡単
- 塩基配列が未知のmRNAの定量ができる
- 発現量の少ないmRNA発現の定量ができる
- 得られる情報量が多い
DNAマイクロアレイ法とRNA-seqのどちらを使うか考える上で確認すべきポイントは、以下の3つです。
- 目的のmRNAの塩基配列はわかっているか?
- サンプルや遺伝子の発現量は十分か?
- サンプルの遺伝子情報の全てが必要か?
- 目的のmRNAの塩基配列が既知(DNAマイクロアレイが使用可能)
- mRNAの発現が十分量ある
- 未知の遺伝子情報を必要としない(目的が既知の遺伝子の定量)
- 塩基配列が未知のmRNAも含めて定量したい
- mRNAの発現量が少ない(または未知)
- 未知の遺伝子情報を探索したい
今回はmRNAの定量方法の一つである、DNAマイクロアレイ法とRNA-Seqについて解説しました。mRNAを定量する他の手法は、下記記事で紹介しています。