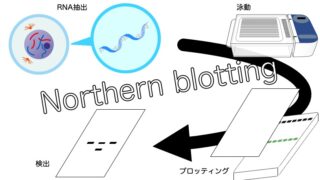
「RNA-Seqについて調べてくるように言われたけど、難しくてよくわからない!」「結局RNA-Seqって何ができるの?」
そんなあなたのために、生命科学系の大学院博士課程を経て、現在も大学の研究現場で務めている私が、RNA-Seqについて解説します。
- RNA-Seqは「mRNAの塩基配列を読んで定量する」手法である
- RNA-Seqでは遺伝子発現を網羅的に定量できる
- RNA-Seqの工程6ステップを解説
- メリットは配列が未知の遺伝子も定量できること
- デメリットはコストとデータ量の多さ
- 遺伝子発現の定量以外に、通常とは違う配列のmRNAの解析にも使われる
RNA-Seqは2000年代に確立された方法で、装置も高額なため、実際にやったことがあるという学生さんは多くないはずです。でも大丈夫!この記事を読むだけで、RNA-Seqを理解するために必要な知識が揃います。
RNA-Seq解析の原理
RNA-Seqとは、遺伝子発現の定量方法の1つです。次世代シーケンサー(NGS)という装置を使って、細胞中の全mRNAの塩基配列を解読します。その結果から、ゲノム上のどの配列がどれくらい転写されているかを解析することで、遺伝子発現を定量します。細胞に含まれる全mRNAの配列を決定するため、特定の遺伝子領域の発現を解析するリアルタイムPCRなどと違い、未知の遺伝子配列であっても解析することができます。
RNA-Seqの工程6ステップ
RNA-Seqは以下のような流れで行います。
- 細胞からmRNAを抽出
- mRNAを断片化
- 2本鎖のDNAを合成
- 両側の末端にアダプターを付ける
- シーケンシング
- マッピング
これらを一つずつ解説していきます。
1. 細胞からmRNAを抽出
まずは、解析したいサンプルからmRNAを抽出します。真核生物の細胞を使う場合は、mRNAの3’末端にあるポリAテールを使って、mRNAのみを精製することができます。具体的には、ビーズなどの担体にT(チミン)が連続したDNAオリゴを結合させ、そこに回収したtotal RNAを加えます。Tが連続したオリゴには、ポリAテールをもつmRNAが結合します。その後担体に結合したものだけを溶出することで、rRNA等が除去されたmRNAサンプルを得ることができます。なお真核生物以外の細胞からmRNAだけを回収する場合には、それぞれの細胞に応じて適切な精製処理を行う必要があります。
2. mRNAを断片化
RNA-Seqでは、短い断片にしたmRNAの配列を次世代シーケンサーで決定し、その後その断片がゲノムのどの領域から転写されたものなのかを解析します。次世代シーケンサーで読む断片の長さは、機械の設定に応じて100bpや150bpとなっており、その設定に合わせてmRNAを断片化します。
次世代シーケンサーでは一般的な長さのmRNA(約500〜2000bp)の塩基配列を断片化せずにそのまま読むことができますが、そうするとコストが高くなってしまいます。
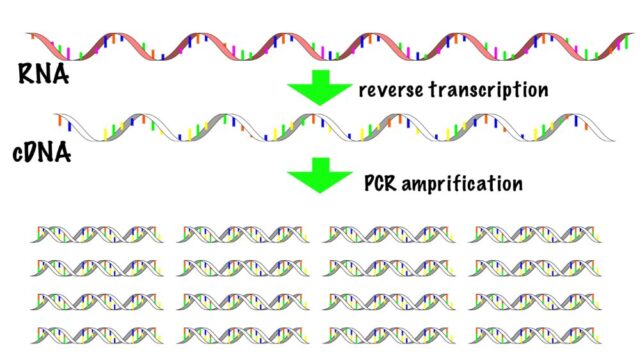
3. 2本鎖のDNAを合成
RNAはとても不安定で分解されやすいため、逆転写によってcDNAを合成します。このcDNAは増幅され、約1000コピーに増やされてからNGSで塩基配列が読まれていきます。
4. 両側の末端にアダプターを付ける
シーケンシングに必要なアダプターを、DNAの両側に付けます。このアダプターには3つの領域が含まれます。
- DNA配列に結合する領域
- シーケンスプライマーが結合する配列
- フローセルに結合する配列
※フローセル:シーケンシングの実施場所となる、レーン付きのガラスのこと。フローセルにはアダプターと結合するためのオリゴヌクレオチドがたくさんあり、この配列とアダプターが持つ相補的な配列が結合する。
5. シーケンシング
つづいて、次世代シーケンサーで塩基配列を解読(シーケンシング)していきます。シーケンシングが終わったら、アダプター配列の部分、解読の精度が低い断片の配列、一定の長さを超えた3’末端側を、データからトリミングします。3’末端側をトリミングするのは、RNA断片の5’側から読み進めるため、3’末端側に近づくにつれ精度が低下していくためです。
6. マッピング
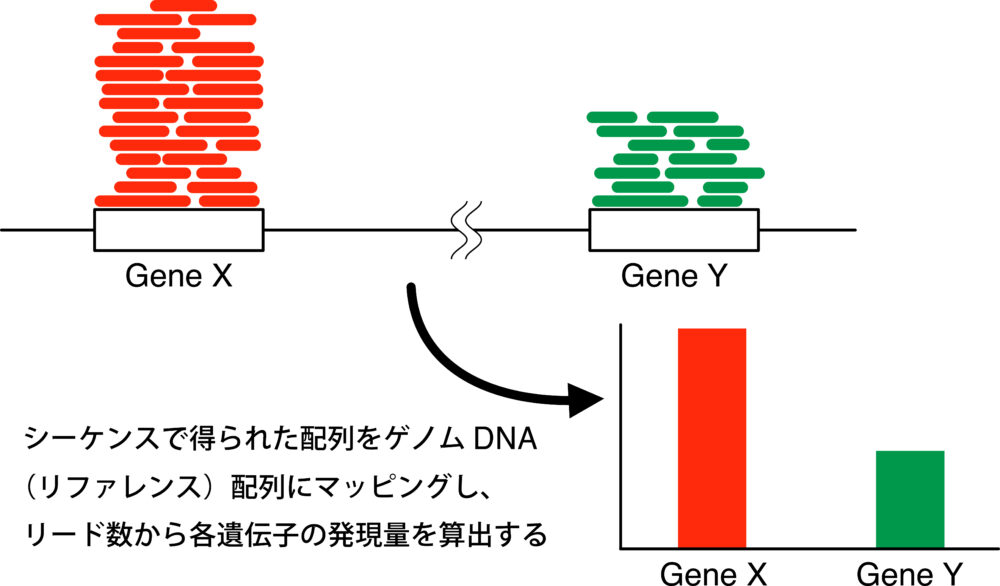
最後に、RNA断片をゲノムDNA配列に当てはめる(マッピング)作業をしていきます。このマッピングによって、どの遺伝子領域が多くmRNAに転写されたか明らかになります。
RNA-Seqのメリット・デメリット
ここまで読むと、「RNA-Seqすごいじゃん!遺伝子発現量を調べられて、塩基配列までわかるんでしょ?」「PCRみたいに一つひとつ調べないで、全部RNA-Seqで解析すればいいのでは?」と思ってしまいそうです。しかし、RNA-Seqにもデメリットはあります。
ここで、遺伝子発現量の定量方法である「リアルタイムPCR」および「DNAマイクロアレイ法」と比較した場合の、RNA-Seqのメリット・デメリットを解説します。
リアルタイムPCRとの比較

リアルタイムPCRは、目的の遺伝子配列のみをRT-PCRで増幅する手法です。その増幅の様子をリアルタイムでモニターすることで、もとのmRNAの定量をすることができます。
リアルタイムPCRと比較したときのメリット
細胞に存在する全てのmRNA配列を網羅的に検出できる
RNA-SeqとリアルタイムPCRとの明確な違いは、「解析する遺伝子領域の違い」です。リアルタイムPCRは特定の領域に絞って解析しますが、RNA-Seqでは細胞に存在する全てのmRNAを解析します。そのため、具体的には以下のことが可能となります。
- 配列が未知のmRNAも定量できる
- 事前に発現しそうな遺伝子領域を予測する必要がない
リアルタイムPCRと比較したときのデメリット
解析に数日~数週間かかる
RNA-Seqには「網羅的な遺伝子発現を解析できる」というメリットがある一方で、解析に時間がかかってしまうというデメリットがあります。
データが膨大になるため、専用サーバーが必要
RNA-Seqを行うと、かなり大きなデータを得ることになります。よって、専用サーバーを準備する必要があります。RNA-Seqで解析を行うためには、次世代シーケンサーを購入する費用はもちろん、専用サーバーも必要となるため、莫大な費用が必要となります。



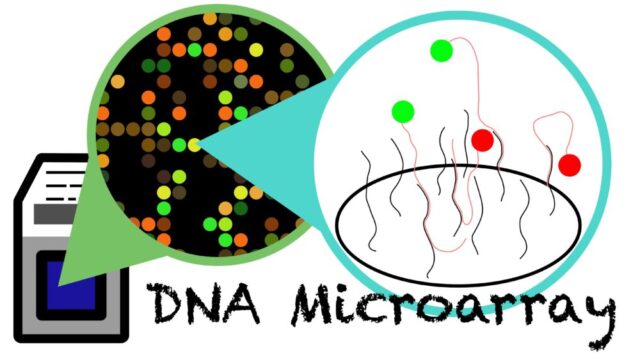
DNAマイクロアレイ法との比較
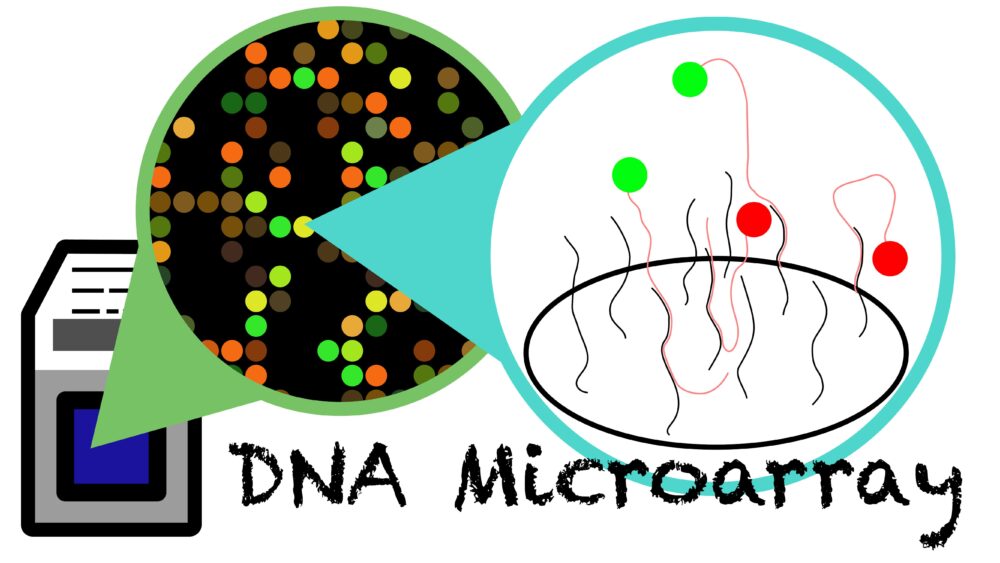

DNAマイクロアレイ法では、数百万の区分(セル)にそれぞれ1種類の1本鎖DNAプローブを固定します。そこにサンプルを加え、ハイブリダイゼーションによって結合したセルを解析することで発現量を解析します。


DNAマイクロアレイ法と比較したときのメリット
未知の遺伝子も検出できる
RNA-seqでは、mRNAの配列を決定していくことで、発現解析を行います。そのため、塩基配列が未知の遺伝子の発現も検出が可能です。一方でDNAマイクロアレイ法での遺伝子発現の定量には、DNAプローブを結合させたDNAマイクロアレイが必要です。DNAマイクロアレイの基盤上のDNAプローブを作成するためには、塩基配列が既知である必要があり、未知の塩基配列には対応できません。
発現量の少ないmRNAの検索性能が高い
RNA-seqでは、原理的に細胞内すべてのmRNAを検出することもできるため、発現量の低いmRNAの網羅的な検索も可能です。一方、DNAマイクロアレイ法では発現量の低いmRNAの定量は困難です。
DNAマイクロアレイ法と比較したときのデメリット
解析データサイズが大きい
RNA-seqでは、1回の解析で数千万〜数億本のmRNA断片の配列を決定し、遺伝子発現の網羅的な解析を行います。そのため、データサイズがギガバイトの単位にもなります。一方、DNAマイクロアレイ法で扱うデータサイズはメガバイト単位と、RNA-seqの1/1,000程度です。そのため、データの保管や解析は、普段使っているPCでも可能です。データサイズが大きい分、DNAマイクロアレイ法よりも解析にかかる費用がかさんでしまいます。
解析にかかる時間が長い
前項で紹介した通り、RNA-seqでは扱うデータサイズがDNAマイクロアレイ法と比較して大きいです。そのため、RNA-seqでは解析に数週間かかる場合もあります。一方DNAマイクロアレイ法は、解析ソフトによっては数分で解析が可能です。



遺伝子発現の定量以外でのRNA-seq活用法
RNA-seqは「未知のmRNA配列を解読できる」という特徴を持つことから、遺伝子発現の定量以外に、以下の解析にも活用されています。
- バリアント
- 融合遺伝子
- 一塩基多型
バリアント
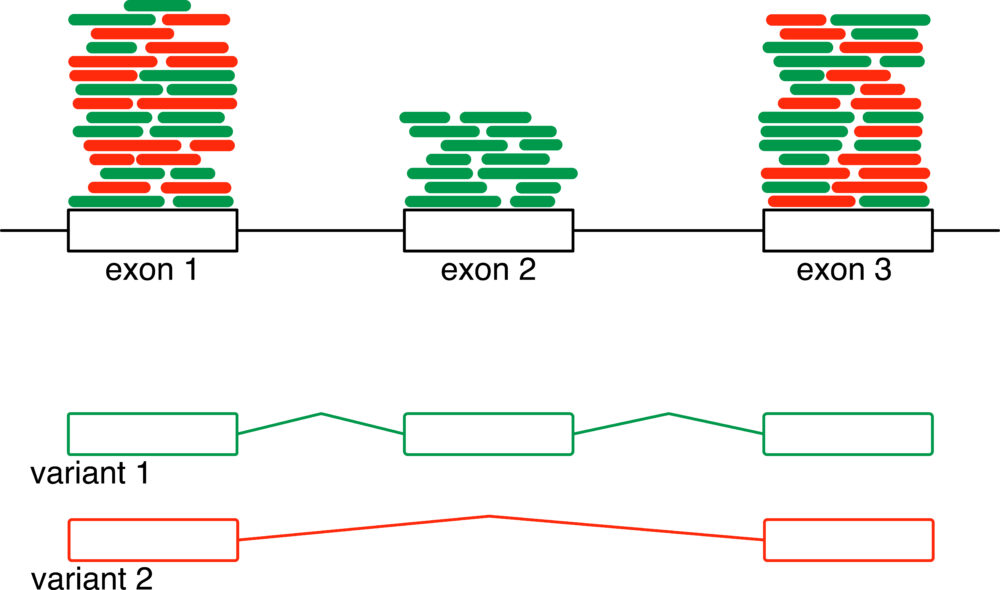

バリアントとは、同じ遺伝子から転写されたにもかかわらず、選択的スプライシングや転写を開始した場所が異なるために生じる、それぞれ異なる転写産物のことです。未だに検出されていないものや特定の条件でのみ生じるものもあるため、リアルタイムPCRのように、このゲノム領域からはこのmRNAが転写されるはずだという予測が必要な解析方法では、見落としてしまいます。一方RNA-seqは、全mRNAの塩基配列を解読するため、こういったバリアントの存在も見つけることができます。
融合遺伝子
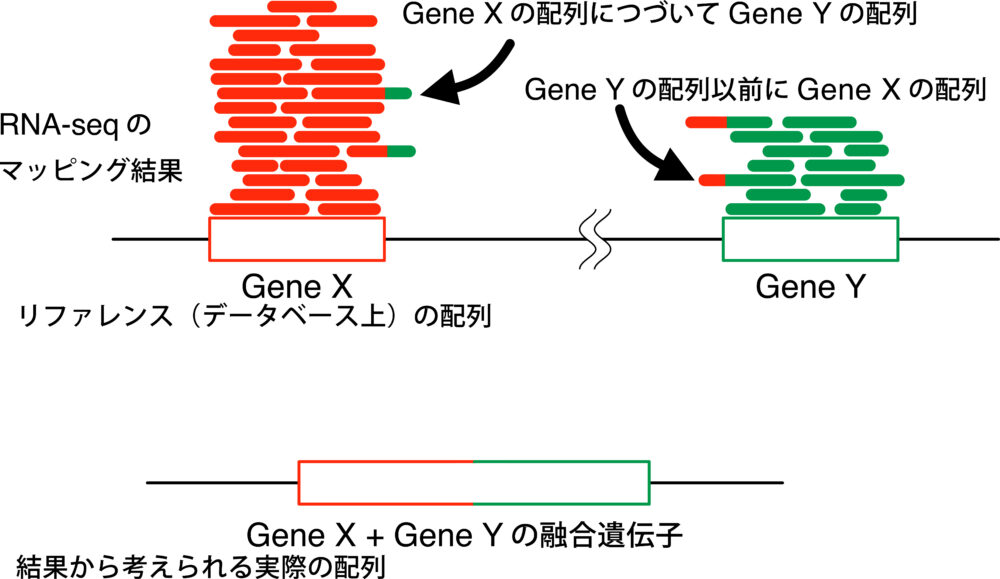

融合遺伝子とは、染色体の転座や欠失、挿入などの変化が起こることで複数の遺伝子の位置が変わってできる遺伝子のことです。この融合遺伝子は疾患や生物の進化に関わっているため、解析することはとても重要です。RNA-seqを用いて全mRNAの全塩基配列を解読することで、こういった融合遺伝子の存在を特定することができます。
一塩基多型
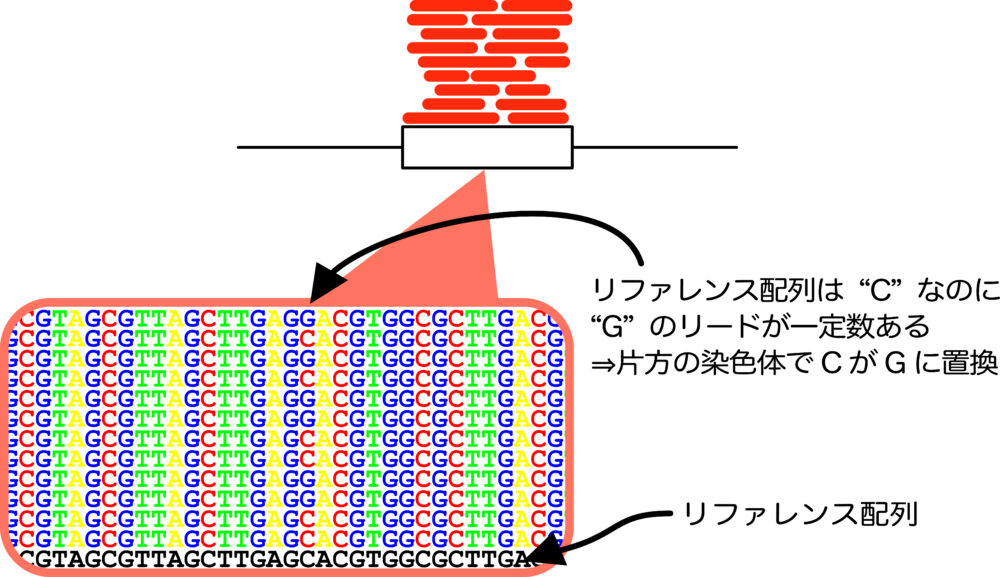

RNA-seqは未知の遺伝子も解析できるという特徴から、1塩基の変化・欠失・挿入の解析にも用いられます。
まとめ
この記事では、RNA-Seqについて解説しました。
RNA-seqは次世代シーケンサーを用いて、サンプル中の全mRNAの塩基配列を決定することで、遺伝子発現を網羅的に定量することができます。
- 細胞からmRNAを抽出
- mRNAを断片化
- 2本鎖のcDNAを合成
- 両側の末端にアダプターをつける
- シーケンシング
- マッピング
また、遺伝子発現の定量以外にも、
- バリアント解析
- 融合遺伝子の解析
- 一塩基多型の解析
遺伝子発現を定量する方法はRNA-seq以外にもあります。他の方法については、下記記事にて紹介しています。