The transcription factor NRF1 (NFE2L1) activates aggrephagy by inducing p62 and GABARAPL1 after proteasome inhibition to maintain proteostasis
Atsushi Hatanaka, Sota Nakada, Gen Matsumoto, Katsuya Satoh, Iori Aketa, Akira Watanabe, Tomoaki Hirakawa, Tadayuki Tsujita, Tsuyoshi Waku, Akira Kobayashi
- 要約
- はじめに
- 1. NRF1はプロテアソームの活性低下を相補してオートファジーを活性化する
- 2. プロテアソーム阻害時にNRF1はオートファジー関連遺伝子の発現を誘導する
- 3. NRF1はプロテアソーム阻害時にオートファジー関連遺伝子の発現を制御する
- 4. NRF1はp62の遺伝子発現を誘導し、aggrephagyを活性化する
- 5. NRF1はプロテアソーム阻害時にp62陽性punctaを形成するために必要である
- 6. NRF1はaggrephagyにおいてGABARAPL1を発現誘導する
- 7. NRF1関連因子NRF2もプロテアソーム阻害時にp62とGABARAPL1の発現を誘導する
- まとめ
- この研究で使っている手法の解説記事
要約
この研究により、NRF1がプロテアソーム活性低下時にaggrephagyを活性化してプロテオスタシスを維持する重要な役割を果たすことが明らかになりました。具体的には、NRF1はプロテアソームが阻害された際に、p62およびGABARAPL1の発現を直接誘導しました。さらに、NRF1はp62陽性punctaの形成と、puncta内でのULK1/TBK1の共局在に不可欠であることが示されました。これに伴い、NRF1はp62のSer403のリン酸化を促進していました。また、NRF1はATG8ファミリーの中で特にGABARAPL1を選択的に誘導し、ユビキチン化タンパク質の除去に関与していました。これらの結果から、NRF1はタンパク質恒常性を維持する上で不可欠な因子であることが示唆されます。
はじめに
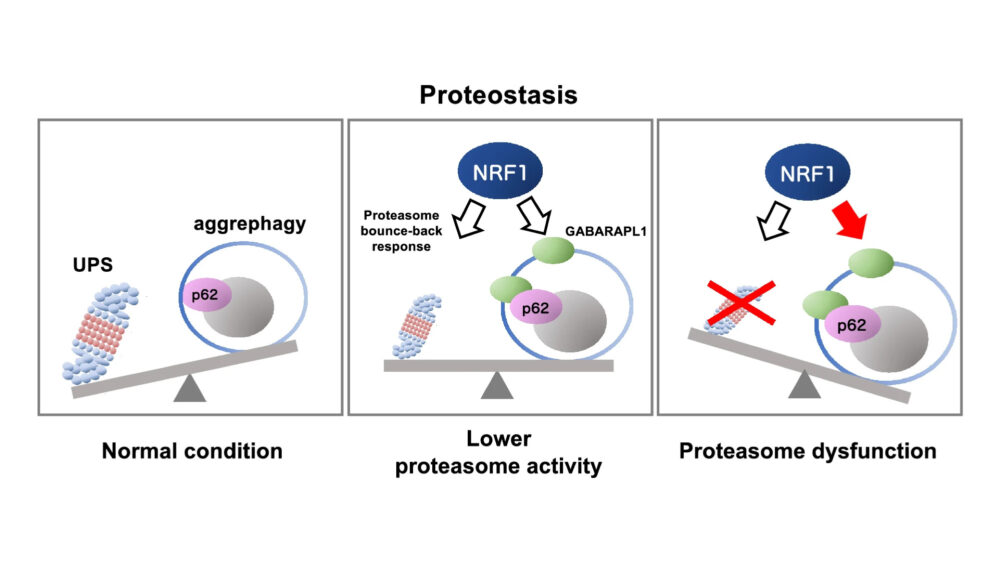
2つのタンパク質品質管理システムの連関
ユビキチン-プロテアソーム(UPS)とオートファジーは、折りたたまれていない・誤って折りたたまれた・損傷したタンパク質によって引き起こされる、細胞機能の深刻な不均衡を防ぎ、タンパク質の恒常性(すなわちプロテオスタシス)を維持するための2つの重要なタンパク質品質管理システムです。UPSおよびオートファジーの機能障害は、プロテオスタシスの不均衡を引き起こし、神経変性疾患、がん、および代謝性疾患をはじめとした老化と関連したヒトの疾患につながります(Dikic I. (2017) Anni. Rev. Biochem.、Deshaies RJ (2014) BMC. Biol.、Lamark T. (2021) Annu. Rev. Cell Dev.)。外部および内部の刺激(たとえば、多発性骨髄腫治療用のボルテゾミブなどの化学的阻害剤、酸化ストレス、異常または過剰なタンパク質含有、または老化など)によって、このシステムのタンパク質分解能力は低下することがあります。プロテアソームの機能が抑制された場合、細胞はオートファジーを活性化してプロテオスタシスを維持し、大きな潜在的に有害なタンパク質集合体を除去します。対照的に、オートファジーの抑制はほとんどプロテアソームを活性化しません。これらの発見は、UPSとオートファジー間にタンパク質品質管理での機能的なコミュニケーションがあることが強力な証拠です。ただし、プロテアソームの阻害後にUPSからオートファジーへの移行を完全に解明するための分子的基盤はまだ明らかにされていません。
転写因子NRF1について
複数のデータから、転写因子NRF1(NFE2L1)がプロテオスタシスにおいて重要な役割を果たしていることが分かっています。NRF1は、NRF2(NFE2L2)およびNRF3(NFE2L3)などとともに、CNC転写ファミリーに属し、プロテアソームが機能不全に陥った後にプロテアソームサブユニット遺伝子の発現を調節することが示されています(Hamazaki J. (2020) Int. J. Mol. Sci.、Sekine H. (2021) Cancers、Ruvkun G. (2023) Cold Spring Harb. Perspect. Boil.)。この細胞内応答は「proteasome bounce-back response」(または「proteasome recovery」とも呼ばれます)。生理的条件下では、NRF1の転写活性は、エンドプラズミック・レチクルム(ER)内での封入と、E3ユビキチンリガーゼHRD1によるER関連タンパク質分解(ERAD)システムにおける分解によって抑制されています(Tsuchiya Y. (2011) Mol. Cell. Biol.、Steffen J. (2010) Mol. Cell、Radhakrishnan SK (2010) Mol. Cell)。プロテアソームの抑制後、NRF1はアスパラギン酸プロテアーゼDDI2による切断を受け、それによりERからの解離と核への移行が起こります(Koizumi S. (2016) Elife、Lehrbach NJ. (2016) Elife)。そして、NRF1は小Mafタンパク質と抗酸化応答要素(ARE)に結合することで、プロテアソーム遺伝子の発現を上昇させ、プロテアソーム活性の回復を促進します。これらの発見を支持する形で、神経特異的なNrf1の欠失は、プロテアソーム活性の低下によりユビキチン化タンパク質が蓄積し、神経変性疾患を伴うマウスを引き起こすことが示されています(Lee CS. (2011) Proc. Natl. Acad. Sci. USA.、Kobayashi A. (2011) Genes Cells)。重要なことに、proteasome bounce-back responseは医学的に重要であり、ボルテゾミブなどによる治療用のプロテアソーム阻害剤によって、癌細胞(多発性骨髄腫細胞など)において迅速にプロテアソーム遺伝子が上昇し、抗がん剤に対する癌細胞の耐性を引き起こすことがあります(Northrop A (2020) Int. J. Mol. Sci.、Gu Y. (2020) Cell. Signal.、Fassmannova D. (2020) Cancers、Chen T. (2022) Blood Adv.、Op M. (2022) Cell Death Dis.、Tomlin FM (2017) ACS. Cent. Sci.)。ただし、プロテアソーム活性が完全に抑制された場合、NRF1がオートファジーなどの他のタンパク質分解システムを強化するかどうかは不明です。
オートファジーの一種aggrephagyについて
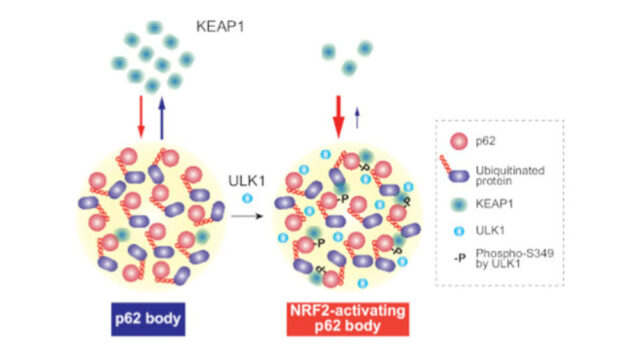
オートファジーは当初、栄養や成長因子の不足時に、タンパク質凝集体や細胞小器官などの細胞内構造物が非選択的かつ大量に分解されるシステムとして同定されました(Dikic I. (2017) Anni. Rev. Biochem.、Lamark T. (2021) Annu. Rev. Cell Dev.)。これらの基質はファゴフォアに取り込まれ、ファゴフォアが閉じられてオートファゴソームが形成され、リソソームと融合した後、オートファゴソームとその中身が分解されます。しかし、最近の研究の進展により、細胞内構造物は選択的にオートファジーによって分解されることが分かってきており、この分解経路は「選択的オートファジー」と呼ばれ、いくつかのストレスやシグナルによって誘導されています(Dikic I. (2017) Anni. Rev. Biochem.)。例えば、プロテアソームの阻害は、タンパク質凝集体を分解する「aggrephagy」と呼ばれる選択的オートファジーを促進します(Kopito RR. (2000) Trends Cell Biol.)。aggrephagyの選択性は、オートファジー受容体の基質に対する結合親和性によって制御されています(Lamark T. (2021) Annu. Rev. Cell Dev.、Johansen T. (2020) J. Mol. Biol.)。これらの受容体の中で、p62タンパク質、またはsequestosome 1(SQSTM1)とも呼ばれるタンパク質は、aggrephagyにおいて2つの重要な役割を果たしています。具体的には、p62陽性のpuncta(セクエストソームとも呼ばれます)の形成と、それらがファゴフォアに取り込まれることです。まず、p62陽性のpunctaは、複数の連続的な段階で形成されます。ubiquitin-associated(UBA)ドメインを介して、p62は蓄積されたUb-タンパク質と結合し、UBAドメイン内のSer407におけるULK1によるリン酸化を促し、UBAドメインを介して形成されるp62の二量体から単量体への遷移と、ULK1、TBK1、またはCK2によるSer403のリン酸化を促進します(Lim J. (2015) PLoS Genet.、Matsumoto G. (2015) Hum. Mol. Genet.、Matsumoto G. (2011) Mol. Cell、Pilli M. (2012) Immunity)。それにより、p62はUb-タンパク質との結合親和性が向上し、その結合によってセクエストソームの相分離形成が引き起こされます(Sun D. (2018) Cell Res.、Turco E. (2019) Mol. Cell、Zaffagnini G. (2018) EMBO J.)。2つ目に、これらのセクエストソームはATG8タンパク質との直接的な相互作用によってファゴフォアに取り込まれ、その後、オートファジー関連のタンパク質分解の対象とされます。これらの知見は、aggrephagyにおけるp62の重要な役割と、ユビキチンがUPSを介した効果的なタンパク質分解だけでなく、aggrephagyにも必要であることを強く示唆しています。
ATG8ファミリー、特にGABARAPL1について
ATG8ファミリータンパク質は、オートファゴソームの形成において重要な分子であり、リン脂質であるホスファチジルエタノールアミン(PE)と共役してオートファゴソームの膜に結合します(Lamark T. (2021) Annu. Rev. Cell Dev.、Jacquet M. (2021) Autophagy、Nieto-Torres JL. (2021) Trends Biochem. Sci.)。哺乳動物において、ATG8ファミリーは7つのオルソログ(LC3A、LC3B、LC3B2、LC3C、GABARAP、GABARAPL1、GABARAPL2)で構成され、アミノ酸配列の類似性に基づいて2つのサブファミリーに分類されています。LC3サブファミリーとGABARAPサブファミリーです。これらのファミリーの機能的な類似性と相違点が報告されていますが、これらの報告を完全に確認するためにはさらなる研究が必要です(Johansen T. (2020) J. Mol. Biol.、Nieto-Torres JL. (2021) Trends Biochem. Sci.)。GABARAPL1と他のATG8ファミリータンパク質は、オートファジー受容体であるp62、NBR1、Optineurin(OPTN)と直接結合し、セクエストソームを形成するオートファゴソームに引き寄せます。さらに、GABARAPL1はATG2とULK1と直接相互作用してオートファゴソームの形成を活性化する役割も報告されています(Grunwald DS. (2020) Autophagy、Bozic M. (2020) EMBO Rep.)。ただし、aggrephagyにおいてGABARAPL1が果たす機能的な役割を完全に特徴づけるには、さらなる詳細な研究が必要です。
本研究の総括
したがって本研究では、プロテアソームの機能障害がaggrephagyを活性化する分子メカニズムを包括的に調べることを試みました。この目標を達成するために、proteasome bounce-back responseの主要な調節因子である転写因子NRF1に焦点を当て、NRF1がこの過渡的なメカニズムを調節することを発見しました。NRF1は、オートファジー関連遺伝子であるp62とGABARAPL1の発現を直接上昇させ、Ub-タンパク質の除去を促進しました。さらに、NRF1がp62陽性のpunctaの形成に重要であることがわかりました。したがって、この研究は、プロテアソームの活性障害によって引き起こされるオートファジーの活性化におけるNRF1の重要な生物学的機能を明らかにするものです。
1. NRF1はプロテアソームの活性低下を相補してオートファジーを活性化する
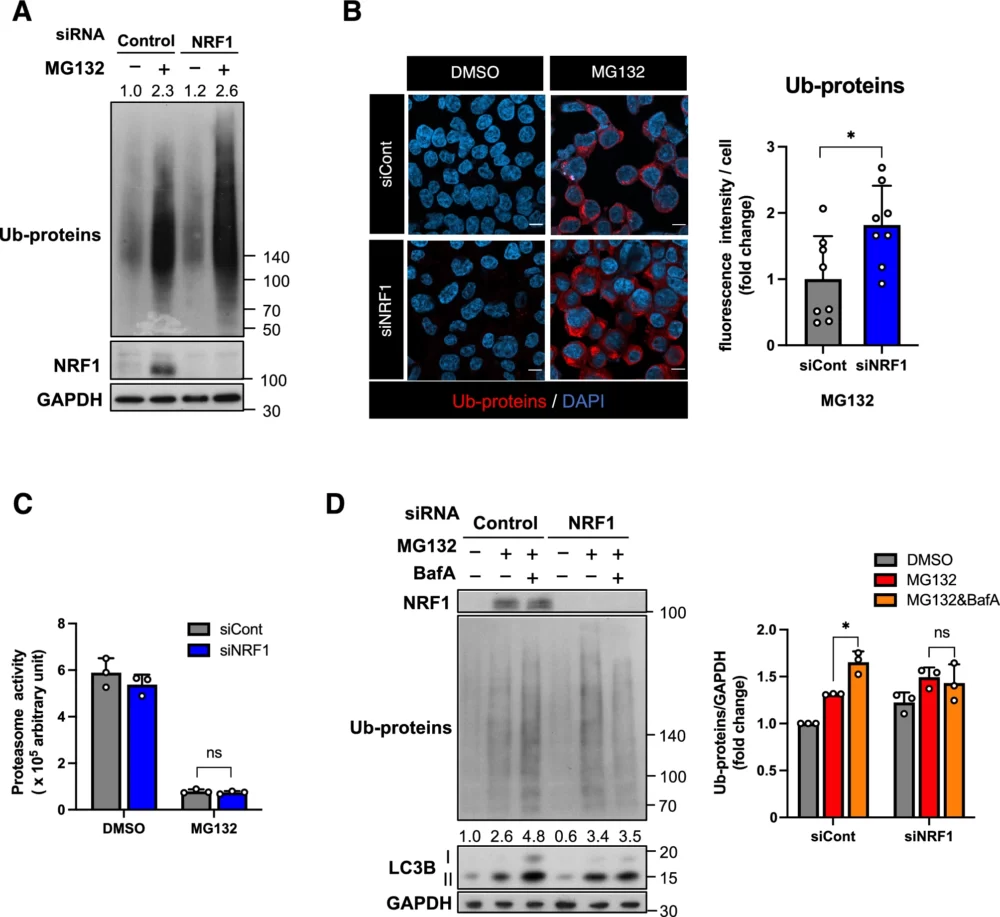
はじめに、プロテアソームの機能が低下したとき、細胞内でオートファジーによるタンパク質分解がNRF1によって亢進される可能性を調べました。ヒト大腸がん由来のHCT116細胞にNRF1に対するsiRNAを導入し、その後、プロテアソームを阻害するMG132を処理しました。これらの細胞から取得した細胞破砕物を用いてウェスタンブロットを行い、MG132処理によってユビキチン化タンパク質が蓄積することを確認しました(図1A)。さらに、NRF1をノックダウンした場合、MG132処理によってユビキチン化タンパク質の蓄積が増加することがわかりました(図1A)。同様に、免疫染色解析でも、MG132処理によって細胞内のユビキチン化タンパク質の量が増加し、NRF1ノックダウンによりこれがさらに増加することが示されました(図1B)。以前の研究から、NRF1はproteasome bounce-back responseを調節していることが示されており(Steffen J. (2010) Mol. Cell、Radhakrishnan SK (2010) Mol. Cell、Tsuchiya Y. (2013) Mol. Cell. Biol.)、NRF1をノックダウンさせた場合、プロテアソームの活性が通常より低下すると考えられました。したがって、これらの実験条件下で、プロテアソームの活性を蛍光基質を用いて測定しました。しかし、予想とは異なり、NRF1ノックダウンはプロテアソームの活性に影響を与えないことが明らかになりました(図1C)。この結果から、NRF1はプロテアソーム以外のタンパク質分解システム、具体的にはオートファジーを促進している可能性があるという仮説を立てました。
上記の仮説を検証するために、プロテアソームの働きを阻害した条件下で、本当にオートファジーがNRF1依存的に促進しているかどうかを調べました。オートファジーにおいて、LC3-IIタンパク質はオートファゴソームの膜に結合し、その後リソソームで分解されます。そのため、オートファジーが活性化されていると、オートファジー阻害剤処理でLC3-IIの量が増えるはずです。具体的には、HCT116細胞にNRF1に対するsiRNAを導入し、それからプロテアソームを阻害するMG132とオートファジーを阻害するbafilomycin A1(BafA)で処理しました。その結果、通常のコントロール条件ではMG132処理によりユビキチン化タンパク質とLC3-IIが増加しました。しかし、NRF1をノックダウンすると、これらの増加が打ち消されたのです(図1D)。これらの結果は、NRF1がプロテアソームの活性低下を補完するためにオートファジーを促進している可能性を強力に支持しています。
2. プロテアソーム阻害時にNRF1はオートファジー関連遺伝子の発現を誘導する
NRF1がオートファジーを促進するのに、どのような遺伝子発現を介して関与しているかを調べるため、HCT116細胞をプロテアソームを妨げる条件下でトランスクリプトーム解析を行いました。具体的には、2つの方法を使用しました。一つは、遺伝子発現を包括的に測定するDNAマイクロアレイ解析、もう一つは、NRF1がゲノム上のどの位置にリクルートされているかを包括的に特定するChIP-seq解析です。
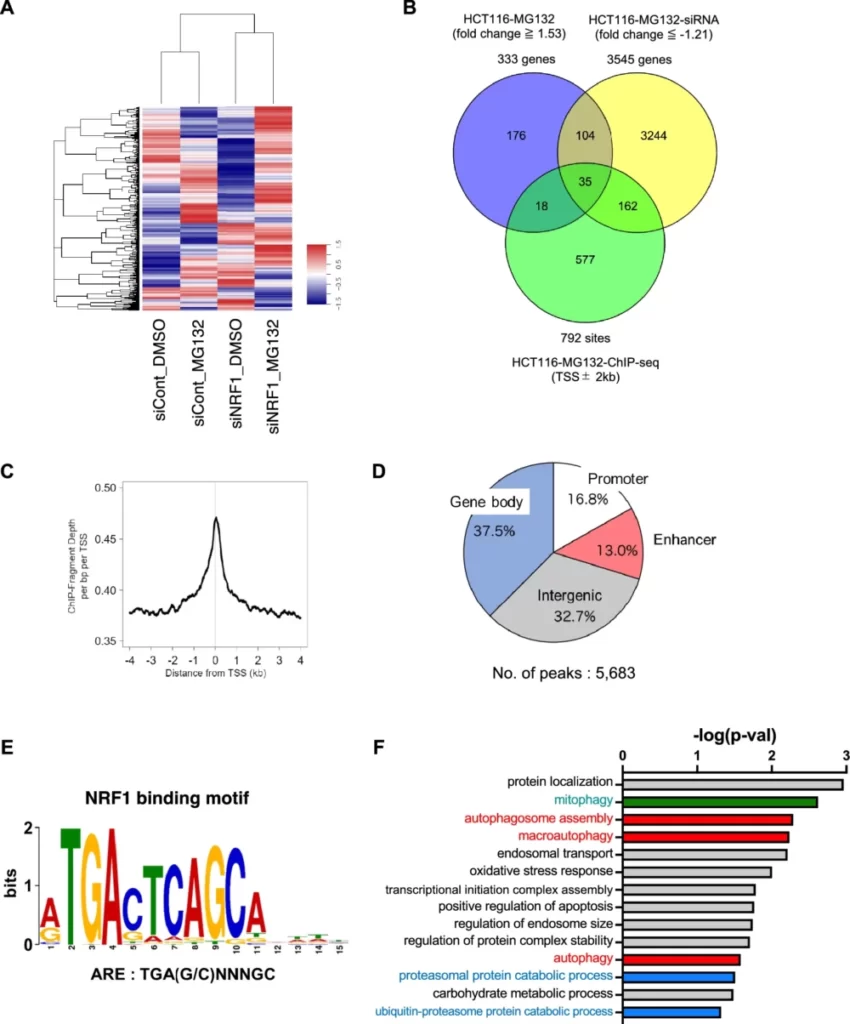
最初に、HCT116細胞を用いてDNAマイクロアレイ解析を実施しました(図2A)。この解析から、プロテアソームの活性が低下した場合、333個の遺伝子が発現が増加することが明らかになりました(fold change ≥ 1.53、図2B)。そして、この333個の遺伝子の中から、139個の遺伝子がNRF1をノックダウンすることで発現が低下することがわかりました(fold change ≤ -1.21、図2B)。言い換えれば、これらの139個の遺伝子は、NRF1の標的遺伝子である可能性が高いということです。
次に、NRF1が結合するゲノム領域を特定するために、MG132処理したHCT116細胞を使用してChIP-seq解析を行いました。この解析には、mouse Nrf1の292-741残基を用いて作製された抗Nrf1ポリクローナル抗体を使用しました(Tsuchiya Y. (2013) Mol. Cell. Biol.)。クロマチン免疫沈降(ChIP)を実施し、得られたDNAサンプルの配列を次世代シーケンス解析で決定しました。
ChIP-seq解析の結果、NRF1は特に転写開始点の近くに強くリクルートされていることが分かりました(図2C)。また、転写開始点から前後2 kbp以内にNRF1が有意にリクルートされている領域は792箇所ありました(図2B)。ゲノム全体を見ると、NRF1結合領域の16.8%がプロモーター領域(転写開始点から前後3 kb以内)、13.0%がエンハンサー領域(転写開始点上流3 kb〜20 kb)、37.5%が遺伝子座中(転写開始点下流3 kb〜転写終結点上流1 kb)、32.7%がintergenic領域(上記以外の領域)に位置していました(図2D)。
さらに、de novoのモチーフ解析ツールであるMEME-ChIP(Machanick and Bailey, 2011)を使用し、これらのNRF1結合領域に抗酸化剤応答配列(TGA(G/C)NNNGC、抗酸化剤応答要素; ARE)が存在することを確認しました(図2E)。このモチーフは、NRF1が認識するコンセンサス配列や、マウスNrf1に関する以前の研究結果と一致しています(Johnsen, Ø. (1996) Nucleic Acids Res.、Johnsen, Ø. (1998) Nucleic Acids Res.、Baird L. (2017) Mol. Cell. Biol.)。したがって、このChIP-seq解析によってNRF1の結合領域を正確に同定できたと結論しました。
DNAマイクロアレイ解析とChIP-seq解析の結果を統合することで、プロテアソーム活性が低下した状態でのNRF1の標的遺伝子候補を35遺伝子を同定しました(図2B)。これらの35遺伝子を用いて、Gene Ontology解析を行いました(DAVID、Huang DW. (2009) Nat. Protoc.)。その結果、これらの遺伝子がNRF1の既知の機能である”positive regulation of proteasomal protein catabolic processes”、”proteasome-mediated ubiquitin-proteasome protein catabolic processes”に関連していることが示されました。さらに、これらの遺伝子が”autophagosome assembly”、”macroautophagy”、”autophagy”といったオートファジー関連の過程にも関与していることが明らかになりました(図2F)。これらの結果から、NRF1がプロテアソーム活性が低下した際にオートファジーを制御していることが高く示唆されます。
3. NRF1はプロテアソーム阻害時にオートファジー関連遺伝子の発現を制御する
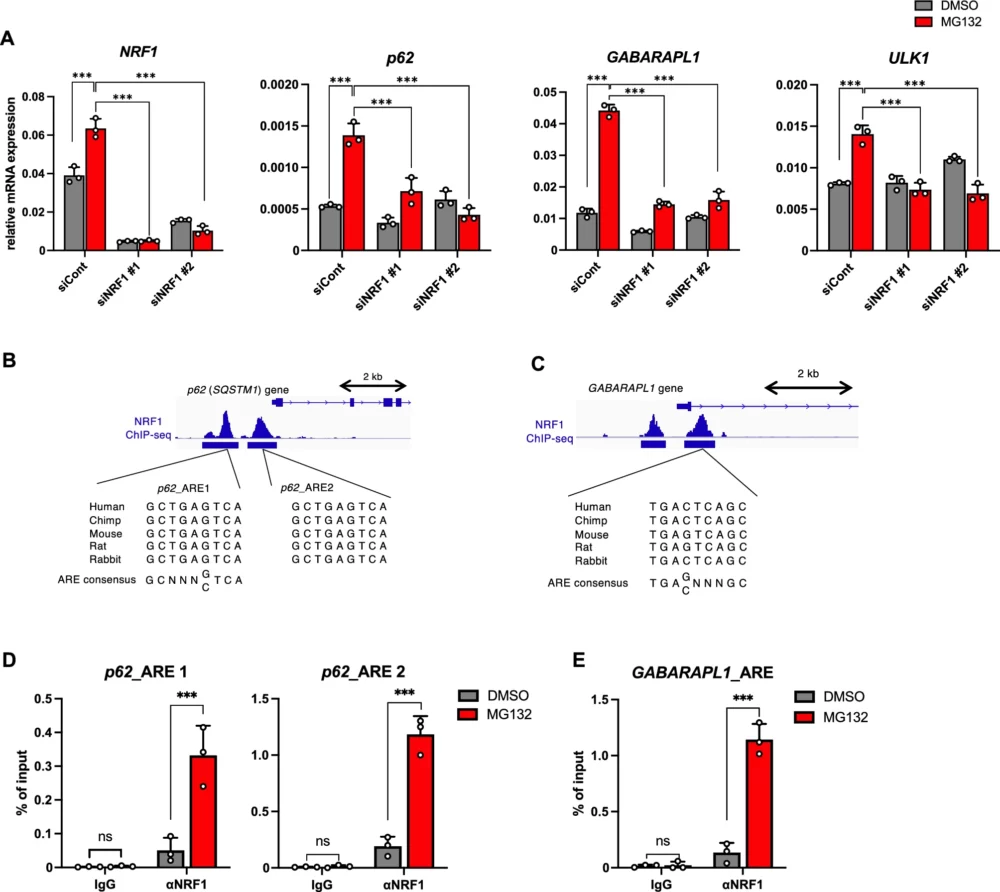
NRF1の標的として同定されたオートファジー関連遺伝子であるp62 Sequestosome 1(SQSTM1)、GABA(A) receptor-associated protein like 1(GABARAPL1)、Unc-51-like autophagy activating kinase 1(ULK1)に焦点を当てて、トランスクリプトーム解析の結果を確認するために、HCT116細胞を使用してRT-qPCR解析とChIP-qPCR解析を実施しました(図3)。
プロテアソームの活性を低下させた際、p62、GABARAPL1、ULK1のmRNA発現が誘導されました。しかし、NRF1をノックダウンすると、プロテアソーム阻害時のp62、GABARAPL1、ULK1の発現が減少したことが明らかになりました(図3A)。また、ChIP-seq解析でNRF1がリクルートされている領域をゲノムデータベースで確認したところ、p62、GABARAPL1、ULK1のプロモーター領域に種間で保存されたARE(抗酸化剤応答要素)が存在することが判明しました。さらに、ChIP-qPCRの結果、これらの領域にNRF1がリクルートされていることが示されました(図3B-E)。
これらの結果から、NRF1がオートファジーの調節メカニズムにおいて重要な役割を果たしていることが強く示唆されます。
4. NRF1はp62の遺伝子発現を誘導し、aggrephagyを活性化する
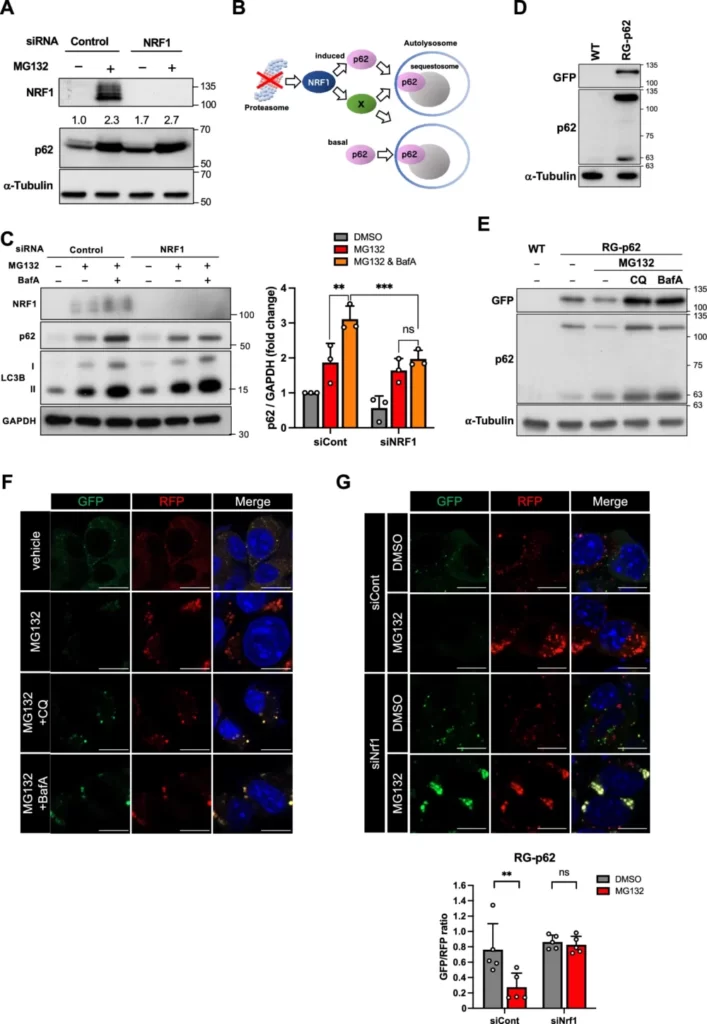
最初に、NRF1-p62経路がタンパク質恒常性にどのような役割を果たすかについて検討しました。NRF1がp62の発現をタンパク質レベルで制御しているかどうかを確認するため、NRF1ノックダウン/プロテアソーム阻害の条件下でウェスタンブロットを実施しました(図4A)。MG132処理によりp62タンパク質の量が顕著に増加したものの、予想外にNRF1ノックダウンではp62タンパク質の減少が確認できなかったばかりか、わずかに増加すらしていました。この予想外の結果を説明するために、”NRF1ノックダウンによりp62の発現だけでなくp62のオートファジーによる分解も抑制される。これらの相反する現象から、p62タンパク質がわずかに増加するという結果になった” という仮説を立てました(図4B)。この仮説を検証するために、オートファジーを阻害するBafA処理がp62タンパク質の量にどのような影響を与えるかを解析しました(図4C)。BafA処理によりLC3-IIの量が顕著に増加し、オートファジーが抑制されていることが示唆されました。この条件下で、p62タンパク質の量も顕著に増加しました。しかし、NRF1をノックダウンした場合、BafA処理によるp62タンパク質の増加は見られませんでした。これらの結果から、プロテアソームが阻害された際、p62がNRF1に依存してオートファジーによる分解を受けていることが示唆されます。
NRF1がaggrephagy(タンパク質凝集体のオートファジー)を介してp62陽性のpuncta(光点状の構造)を分解するかどうかを評価するために、細胞内でのp62タンパク質の分解をモニターするために、RG-p62というレポータータンパク質を開発しました。このレポータータンパク質は、全長のp62に赤色蛍光タンパク質(red fluorescent protein; RFP)と緑色蛍光タンパク質(green fluorescent protein; GFP)を結合させたもので、オートファジー活性を評価するためのシステム(Kimura S. (2007) Autophagy)を応用したものです。
具体的には、RFPは中性の細胞質と酸性のリソソーム内の両方で発光できますが、GFPは中性の細胞質でのみ発光します。したがって、オートファジーによるp62タンパク質の分解が活性化している条件下でRG-p62を発現した細胞を観察すると、RFPのみが発光する輝点が多く見られます。このRG-p62レポータータンパク質を安定的に発現させたNeuro2a細胞を用いて、実験を行いました。
まず、RG-p62が発現していることを確認するために、野生型の細胞株との比較を行いました。GFPおよびp62に対する抗体を使用してウェスタンブロッティングを行った結果、野生型には見られない分子量約115kDaのバンドがRG-p62発現細胞で検出されました(図4D)。このバンドは、RG-p62タンパク質の想定分子量と一致しています。また、RG-p62を発現させたNeuro2a細胞をプロテアソーム阻害剤MG132で処理すると、RG-p62タンパク質量が減少しました。さらに、クロロキン(chloroquine; CQ)もしくはBafAとの共処理により、RG-p62タンパク質の蓄積が確認されました(図4E)。
次に、Neuro2a細胞におけるRG-p62によるpunctaの形成を解析しました。通常の培養条件下では、RG-p62レポーターは細胞質に小さなpunctaを形成し、GFPとRFPの蛍光が両方検出されました(図4F)。しかし、MG132処理によりプロテアソームが阻害され、aggrephagyが活性化すると、このレポーターは大きな構造体を形成し、RFPの蛍光のみが検出されました。この結果は、punctaがリソソームと融合し、リソソーム内の酸性環境によりRG-p62のGFP蛍光が消失していることを示唆しています。さらに、CQもしくはBafAとの共処理で、RG-p62レポーターのGFP蛍光が回復することが確認されました。
これらの結果から、RG-p62レポーターがp62依存的なaggrephagyによるタンパク質分解を再現できており、リソソームでのタンパク質分解が行われていることが示唆されます。
その後、このp62レポーターを使用してNrf1ノックダウンがaggrephagyによるp62の分解に及ぼす影響を、細胞内のGFP/RFPの蛍光比を定量することで検証しました(図4G)。図4Cの結果と一致して、Nrf1をノックダウンするとプロテアソーム阻害によるGFP/RFP比の減少は観察されませんでした。つまり、Nrf1はp62 punctaのオートファジーによる分解を促進していることが確認されました。これらのデータから、プロテアソーム活性が低下した際、NRF1はp62遺伝子の転写誘導だけでなく、aggrephagyによるp62陽性punctaの分解も活性化していることが示唆されました。
5. NRF1はプロテアソーム阻害時にp62陽性punctaを形成するために必要である
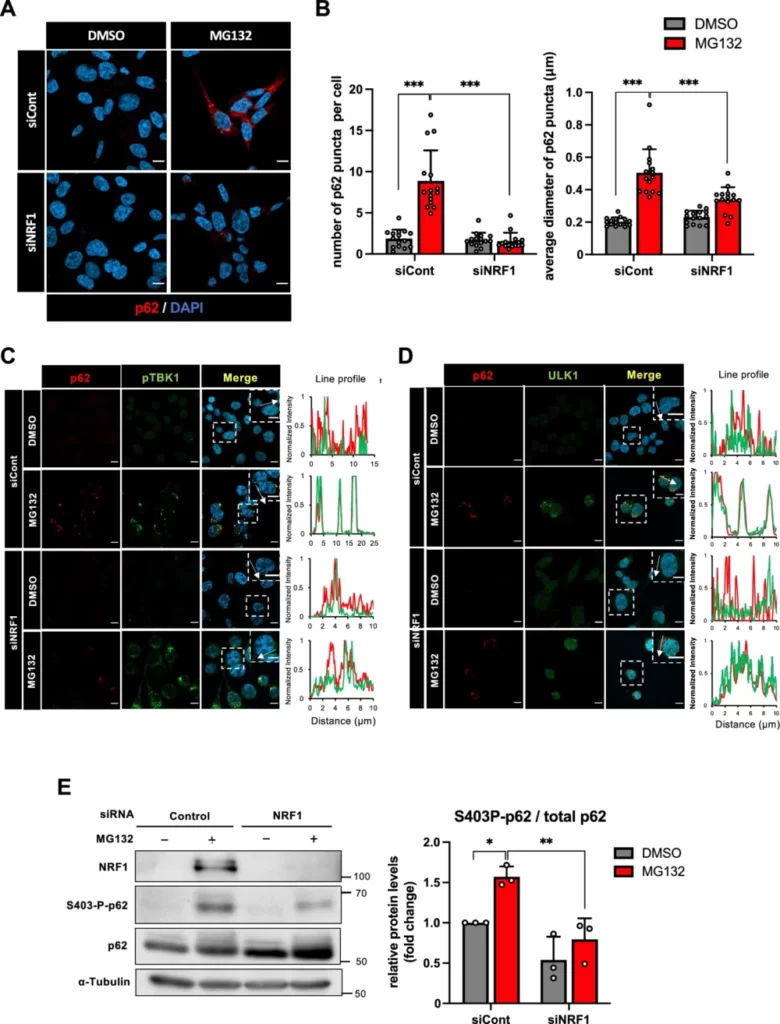
これまでの研究から、プロテアソーム阻害がp62陽性puncta(光点状の構造)の形成を増加させることが知られています(Nakaso K. (2004) Brain Res.、Dikic I. (2017) Anni. Rev. Biochem.、Lamark T. (2021) Annu. Rev. Cell Dev.)。したがって、プロテアソーム阻害によるp62陽性puncta形成にNRF1が関与しているかどうかを検討しました。MG132処理により増加するp62陽性punctaは、NRF1ノックダウンによりpunctaの数と大きさが減少することが観察されました(図5A-B)。図4Aおよび図4Cで示したように、NRF1をノックダウンしても細胞内にはp62タンパク質は存在しています。しかし、この条件下ではpuncta形成が行われなかったのです。これらの結果から、NRF1がプロテアソームの活性低下によるp62陽性puncta形成においても重要な役割を果たしていることが示唆されました。
p62は、TBK1やULK1などの多くのリン酸化酵素によってリン酸化され、相分離によるpunctaを形成します(Matsumoto G. (2015) Hum. Mol. Genet.、Matsumoto G. (2011) Mol. Cell、Pilli M. (2012) Immunity、Sun D. (2018) Cell Res.、Turco E. (2019) Mol. Cell、Zaffagnini G. (2018) EMBO J.)。NRF1によるp62陽性puncta形成の分子メカニズムを解明するため、p62がリン酸化TBK1/ULK1と共局在するかどうかを調査しました。免疫染色の結果、p62とリン酸化TBK1、p62とULK1はpuncta内で共局在し、NRF1ノックダウンによりp62陽性punctaが消失すると、この共局在も消失することが示されました(図5C-D)。これらの結果から、ULK1およびTBK1によってp62のSer403がリン酸化され、punctaを形成している可能性が考えられます。この仮説を確かめるため、NRF1ノックダウンがp62のリン酸化に及ぼす影響を検討しました。その結果、NRF1ノックダウンによりSer403がリン酸化されたp62の量が減少することが示されました(図5E)。これらの結果から、プロテアソーム阻害によるp62陽性puncta形成には、NRF1依存的なp62のリン酸化が必要である可能性が見出されました。
6. NRF1はaggrephagyにおいてGABARAPL1を発現誘導する
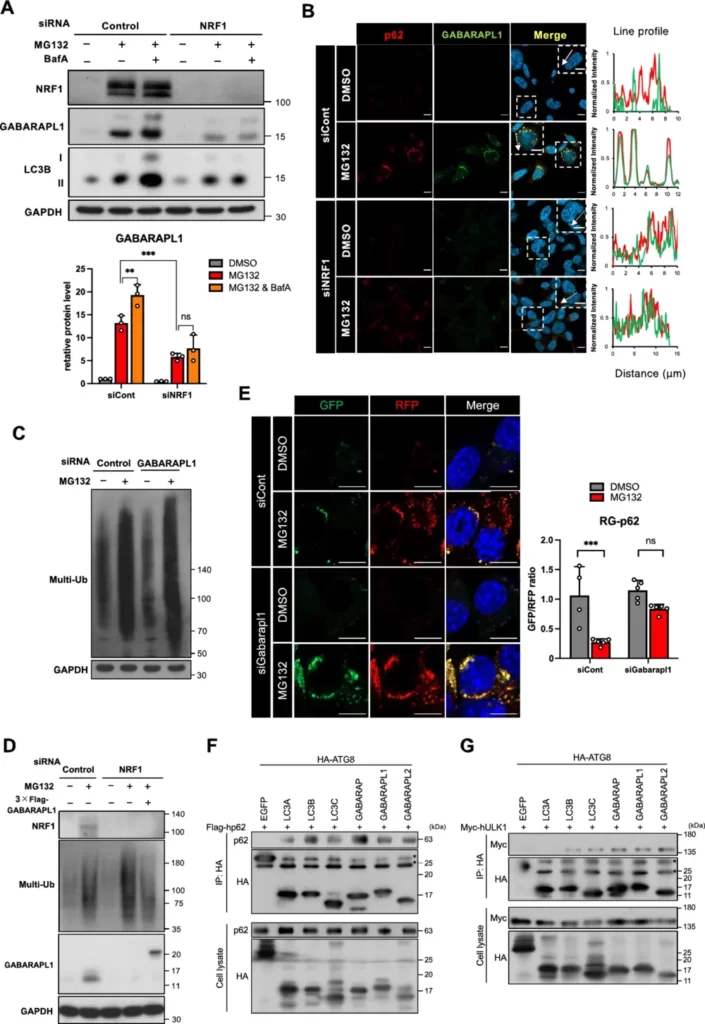
次に、NRF1がGABARAPL1タンパク質量を制御できるかどうかを検討しました。p62と同様に、MG132処理によりGABARAPL1の量が増加し、さらにBafA共処理でその増加が強化されました(図6A)。そして、MG132処理によって増加したGABARAPL1タンパク質の量は、NRF1ノックダウンによって顕著に減少したことが示されました。つまり、GABARAPL1タンパク質はNRF1によってその量が制御されていることが示唆されました。次に、免疫染色を使用して、MG132処理でGABARAPL1とp62陽性punctaが共局在していることを確認しました。さらに、NRF1ノックダウンによってp62陽性punctaが消失すると、共局在も消失することが観察されました(図6B)。これらの結果から、プロテアソーム阻害によってaggrephagyに寄与するGABARAPL1タンパク質の増加が引き起こされ、さらにこの増加がNRF1に依存していることが示唆されました。
さらに、プロテアソーム阻害によって活性化されるNRF1-GABARAPL1経路がaggrephagyによってユビキチン化タンパク質を除去する役割を検証するために、GABARAPL1をノックダウンしMG132処理した際のユビキチン化タンパク質の量を調査しました。その結果、GABARAPL1をノックダウンすると、ユビキチン化タンパク質の量が増加することが確認されました(図6C)。さらに、NRF1をノックダウンしてユビキチン化タンパク質の除去が減少する現象は、GABARAPL1を過剰発現させることでレスキューされました(図6D)。また、RG-p62レポーターを使用して、プロテアソーム阻害によってp62陽性punctaがオートファジーによって分解されるプロセスにおいて、Gabarapl1をノックダウンしてもp62陽性punctaが減少しなかったことも確認されました(図6E)。これらの結果から、タンパク質の恒常性を維持するメカニズムにおいて、プロテアソーム阻害時にNRF1-GABARAPL1経路がaggrephagyを介して重要な役割を果たしていることが示唆されます。
NRF1が選択的にGABARAPL1を誘導する生化学的な意義を理解するために、GABARAPL1を含むATG8ファミリーとp62/ULK1との結合アフィニティを免疫沈降で調査しました。その結果、p62およびULK1はGABARAPL1だけでなく、他のATG8ファミリーとも相互作用することが確認されました(図6F-G)。ATG8ファミリーの中で特にGABARAPL1とのアフィニティが高いという証拠は得られませんでした。この結果から、NRF1-GABARAPL1経路がaggrephagyで効果を発揮するためには、NRF1によってGABARAPL1が遺伝子発現レベルで量的に増加させられることが重要であることが示唆されます。
7. NRF1関連因子NRF2もプロテアソーム阻害時にp62とGABARAPL1の発現を誘導する
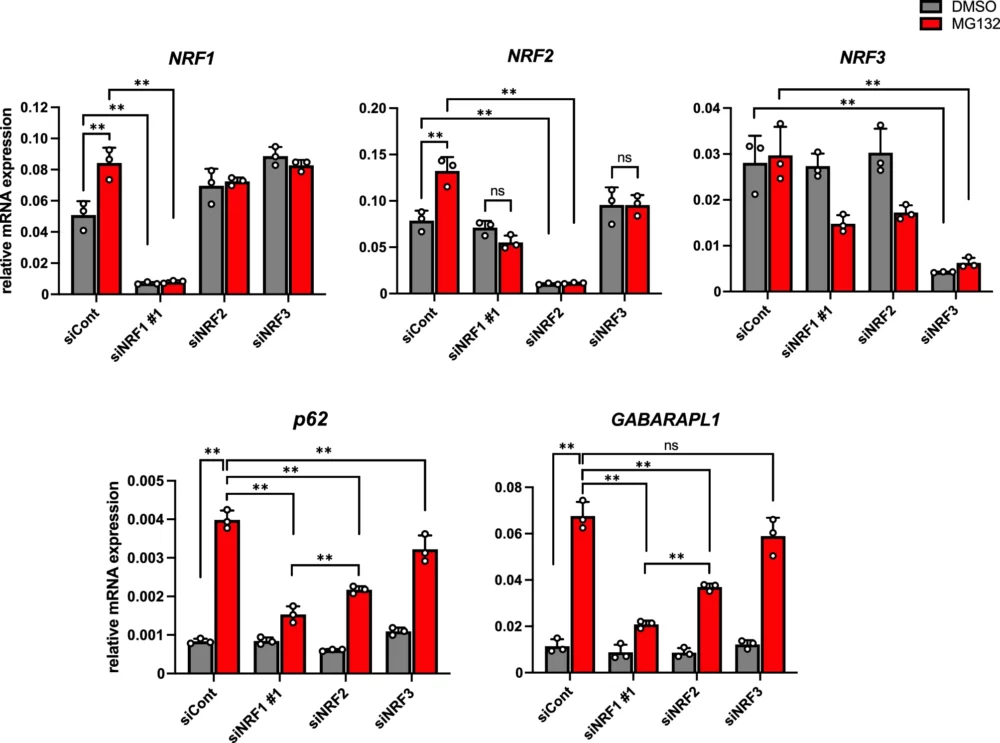
NRF2がp62を誘導してオートファジーを制御していることが以前の研究で示唆されています(Sanchez-Martin P. (2018) J. Cell Sci.)。そこで、NRF1関連因子であるNRF2およびNRF3がプロテアソーム阻害時のaggrephagy活性化に関与しているかどうかを調査しました。このために、NRF2およびNRF3に対するsiRNAを用いてp62およびGABARAPL1の発現を検討しました(図7)。結果として、NRF2をノックダウンすると、MG132処理によって誘導されるp62とGABARAPL1の発現が減少することが確認されました。ただし、NRF1ノックダウンに比べて減少割合は小さかったです。興味深いことに、NRF1はNRF2遺伝子の発現を活性化していることも示唆され、NRF1がNRF2を介してp62およびGABARAPL1の発現を誘導している可能性が示唆されました。一方、NRF3のこれらの遺伝子の発現制御への関与は弱かったです。この結果から、aggrephagyにおいてはNRF1とNRF2が協力して、p62とGABARAPL1の遺伝子発現制御を行っていることが示唆されます。
まとめ
この研究により、NRF1がプロテアソーム活性低下時にaggrephagyを活性化してプロテオスタシスを維持する重要な役割を果たすことが明らかになりました。具体的には、NRF1はプロテアソームが阻害された際に、p62およびGABARAPL1の発現を直接誘導しました。さらに、NRF1はp62陽性punctaの形成と、puncta内でのULK1/TBK1の共局在に不可欠であることが示されました。これに伴い、NRF1はp62のSer403のリン酸化を促進していました。また、NRF1はATG8ファミリーの中で特にGABARAPL1を選択的に誘導し、ユビキチン化タンパク質の除去に関与していました。これらの結果から、NRF1はタンパク質恒常性を維持する上で不可欠な因子であることが示唆されます。
この研究で使っている手法の解説記事